La pandémie de COVID-19 fait des milliers de morts et l'incidence mondiale est mesurée en centaines de milliers. Des antiviraux et des vaccins sont recherchés de toute urgence pour contrer cette menace croissante. Maintenant, une nouvelle étude de la dynamique structurale de la protéine de pointe SARS-CoV-2 publiée dans le serveur de préimpression bioRxiv * en juillet 2020, un rapport sur les épitopes qui pourraient être des cibles immunitaires possibles pour le développement de vaccins.
Sommaire
SRAS-CoV-2 et glycosylation
Le virus est un virus enveloppé avec une protéine de pointe trimérique à sa surface qui est responsable de sa liaison au récepteur de l'enzyme de conversion de l'angiotensine (ACE) 2. Cela fait passer le commutateur conformationnel à un état de post-fusion, où la membrane virale fusionne avec la membrane de la cellule hôte et le virus pénètre dans la cellule hôte.
Les résidus de glycane abondants de la protéine de pointe empêchent les anticorps de la cibler et entravent également l'accès aux médicaments. Par conséquent, la présente étude visait à capturer les changements conformationnels de la protéine Spike (S) et le changement dynamique du revêtement de glycane. Les chercheurs ont utilisé des simulations de dynamique moléculaire (MD) pour brosser un tableau vivant de la protéine S glycosylée et identifier des épitopes immunogènes candidats, en particulier dans la sous-unité S2, qui est responsable de la fusion du virus avec la cellule hôte.

Vue du modèle atomistique simulé contenant quatre protéines S glycosylées et ancrées dans une membrane dans une boîte de simulation hexagonale. Trois protéines sont représentées en surface, les glycanes étant représentés par des bâtonnets verts. Une protéine est représentée dans une représentation de bande dessinée, avec les trois chaînes colorées individuellement et les glycanes omis pour plus de clarté. L'eau (transparente) n'est indiquée que pour la moitié inférieure et arrière de la boîte hexagonale, et les ions sont omis pour plus de clarté.
Accessibilité de Spike Protein
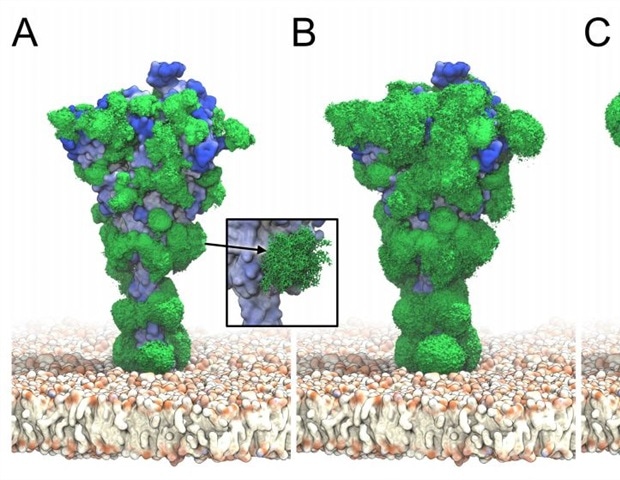
Les anticorps doivent avoir accès à des épitopes pour s'y lier. L'analyse actuelle a montré que le bouclier dense des glycanes réduisait considérablement l'accessibilité de la protéine S aux anticorps. À un moment donné, seule une petite partie de la surface de la pointe est réellement couverte par ces résidus de sucre, mais ils sont si dynamiques qu'ils protègent efficacement toute la surface. Cela a été vu à la fois par l'analyse des rayons et les études d'amarrage Fab.
Les effets de blindage les plus importants sont observés dans la partie la plus proche de la membrane, où la bobine HR2 non glyquée est presque entièrement ouverte à l'attaque des anticorps. Pourtant, avec la glycosylation, il devient inaccessible au fragment Fab et à d'autres grosses molécules.

Dynamique du glycane S à partir de simulations MD µ 2 µs. Les isosurfaces à densité d'électrons de glycane moyennées sont montrées respectivement à des niveaux de contour élevés (A), moyens (B) et bas (C). La surface des protéines bleu à blanc indique une accessibilité élevée à faible dans l'analyse des rayons. (Encart) Instantanés (bâtons) d'un glycane biantenné, à noyau fucosylé et sialylé à la position 1098 le long de la trajectoire MD.
Rigidité des protéines de pointe
La protéine S s'est avérée avoir à la fois des régions rigides ou structurées, qui se lient fortement et spécifiquement à l'anticorps, et des régions dynamiques. Le domaine de liaison aux récepteurs (RBD) et les régions voisines sont tout aussi flexibles, ce qui soutient la recherche montrant des différences marquées dans la structure de la chaîne peptidique à la fois dans les états ouvert et fermé.
D'un autre côté, le domaine S2 qui couvre le peptide de fusion a des structures protéiques rigides, qui peuvent être nécessaires pour maintenir cette partie de la molécule stable dans la configuration de préfusion.

Épitopes identifiés à partir de simulations MD et d'analyses bioinformatiques. Les scores d'accessibilité de l'analyse des rayons (A) et de l'amarrage du corps rigide Fab (B) sont combinés avec les scores de rigidité (C), tous calculés en moyenne sur 4 × 1,93 µs de simulations MD de protéine S. Sont également inclus (D) un score de conservation de séquence (31), et (E) la prédiction de signature de séquence d'épitope BepiPred-2.0. (F) Score d'épitope combiné. (G) Sites de liaison des anticorps neutralisants connus. Une intensité de couleur plus élevée dans A-F indique un score plus élevé et une intensité de couleur plus élevée dans G indique des sites se liant à plusieurs anticorps différents.
Conservation des protéines de pointe
Les chercheurs soulignent l'importance de développer des vaccins qui provoquent des anticorps contre des épitopes relativement stables pour empêcher l'émergence de mutations d'échappement et permettre aux nouvelles souches de se répliquer et de devenir prédominantes. L'analyse actuelle a révélé que la protéine S est hautement conservée et aucune variante n'est présente dans les grandes bases de données actuelles pour plus de la moitié des acides aminés de cette protéine.
Prédire l'immunogénicité
Les chercheurs soulignent que «les régions conservées, rigides et accessibles présentent de bons candidats pour la liaison des partenaires protéiques en général.» En utilisant ces caractéristiques, ils ont trouvé 9 épitopes candidats, dont 4 sont connus et les nouveaux épitopes restants. Tous se trouvaient dans la région de tête de la protéine de pointe, une partie structurée de la molécule, contrairement à la région de tige flexible et inaccessible.
Accord avec des recherches antérieures
Une précédente équipe de chercheurs a découvert que l'anticorps neutralisant CR3022 se liait à l'ectodomaine de la protéine S, à un épitope au-delà de la région de liaison ACE2. Pour réaliser cette liaison, au moins deux des trois protomères S doivent être en configuration ouverte. Cet épitope est récupéré dans l'analyse actuelle, ainsi que les épitopes qui se lient à d'autres anticorps comme CB6, H104 et S309.
Deux des autres épitopes correspondent également aux sites de liaison RBD déjà signalés pour les anticorps neutralisants, montrant, selon les mots des chercheurs, « que notre méthodologie d'identification des épitopes est robuste. »
Des sites de glycosylation dévoilés
L'étude a également confirmé et poursuivi des recherches antérieures sur l'impact de différents modèles de glycosylation sur l'accessibilité de la protéine. Ils ont constaté que, que le mannose seul soit inclus ou que le bouclier glycane complet soit pris en compte, Fab était bloqué efficacement de 60% et 80%, respectivement.
Les chercheurs disent que « même une couverture légère en glycane réduit fortement l'accessibilité aux anticorps de la protéine ».
Caractéristiques des épitopes immunogènes
Ils ont constaté que les trois premiers épitopes avaient des boucles flexibles ainsi que des brins pliés bêta. La suivante était sur une hélice alpha flanquée d'une autre, allongée sur une feuille bêta de cinq brins, un arrangement très stable.
Les deux suivants sont sur la partie apicale du RBD sur la protéine S, principalement des boucles flexibles qui montrent des changements de conformation dans les états ouvert et fermé. D'autres se composent de boucles flexibles longues et courtes ou d'une hélice stable.
Conception future du vaccin
Les chercheurs ont cherché à présenter les épitopes identifiés pour maintenir un profil immunogène robuste. Ils disent que puisque la protéine S est composée de domaines distincts si ces domaines se replient indépendamment, ils pourraient conserver les épitopes et ainsi permettre aux anticorps d'être déclenchés par un vaccin. Cela pourrait peut-être être servi en modifiant convenablement les séquences.
L'étude conclut: «L'approche que nous avons introduite dans cet article pourrait être étendue pour prédire les épitopes à partir d'une analyse intégrée de divers bêtacoronavirus, dans le but ultime de produire un vaccin universel garantissant une large protection contre toute la famille des virus.»
*Avis important
bioRxiv publie des rapports scientifiques préliminaires qui ne sont pas évalués par des pairs et, par conséquent, ne doivent pas être considérés comme concluants, orienter la pratique clinique / les comportements liés à la santé, ou traités comme des informations établies.